Alineamiento Angular#
Stmani3 divide el alineamiento en dos etapas:
-
Alineamiento Angular: Corrige las desviaciones de apuntamiento de las cámaras: Ángulos de Perspectiva Horizontal y Vertical, Rotación sobre el eje óptico y desviaciones de la Longitud Focal nominal. Define también uno de los puntos homólogos como Datum, a partir del cual se sitúan los márgenes de la foto alineada.
El A. Angular proporciona los parámetros para transformar las imágenes originales en las que tomarían dos cámaras ideales con los ejes ópticos paralelos y los sensores alineados. Pero esta etapa no genera imágenes de salida, solo genera un fichero "side-car" *.pair que contiene los parámetros anteriores.
Todas estas operaciones se realizan en la pestaña "Ajuste Angular" de la zona de visionado -
Alineamiento Horizontal y Renderizado: Se basa en aplicar prescripciones de tamaño y de disparidad, con las que se genera el par de imágenes de salida en varios formatos posibles.
Estas prescripciones no afectan al Alineamiento Angular (ver Calidad de Alineamiento), aunque sí a las indicaciones de Calidad. Por eso conviene definir las prescripciones durante el Ajuste Angular usando el botón "Previsual.Render":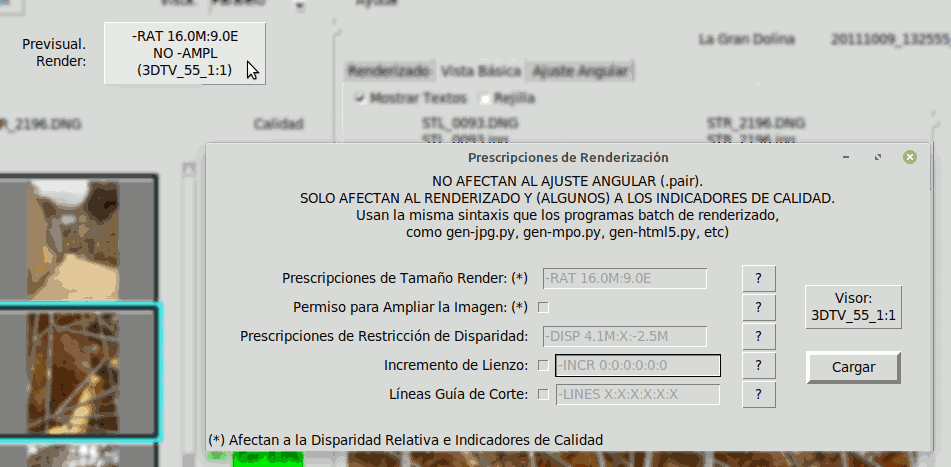
Ver Renderizado para más detalles
Elementos de Alineamiento#
Puntos Homólogos#
El alineamiento angular está basado en un cálculo matemático a partir de las posiciones de un conjunto de puntos homólogos.
Es importante que estos puntos estén bien determinados y que cubran aproximadamente todas las zonas de las imágenes en extensión y en profundidad.
Uno de los puntos se define como Datum. Es el origen de los márgenes en la imagen alineada angularmente, y también es la referencia de disparidad para el resto de puntos homólogos. Debido a esto el Datum tiene cero disparidad horizontal y vertical. Aunque el usuario puede fijar un valor de disparidad horizontal distinto de cero (ver Profundidad del Datum)
Se necesita una cierta cantidad de puntos. El mínimo estricto sería el número de parámetros a resolver más 1 (ver Parámetros de Transformación Angular), que en el caso máximo sería 9. Es frecuente realizar alineamientos con parámetros reducidos, lo que posibilita teóricamente alinear con solo 3 ó 4 puntos, pero eso es más propenso a errores de disparidad vertical aunque solo sea por no cubrir bien todas las zonas de la imagen. Si es posible se aconseja tener bastante más de 9 puntos.
Alineamiento Automático#
La opción de grupo "AUTO AJUSTAR" realiza una búsqueda automática de puntos homólogos, con los que realiza el alineamiento automático del par de imágenes. Hay dos formas de iniciar un alineamiento automático:
- Grupo de pares marcados, que pueden ser todos los del directorio si se desea (ver Opciones de Grupo en manejo de miniaturas). Solo se puede realizar en modo "Emparejados", y salva los resultados en los ficheros .pair correspondientes.
- Cualquier par de imágenes seleccionado (ver Opciones de par individual). Se puede realizar en modo "Emparejados" o "Aleatorio". No salva el resultado y advierte al operador para que lo salve si desea.
Ajustes Anteriores#
En caso de que la imagen tuviera un ajuste previo, se aplican los siguientes criterios:
- Párametros Angulares: Se utilizan como valores iniciales para las iteraciones del nuevo ajuste (ver aproximaciones sucesivas)
- Puntos H. anteriores: No se mantienen, pero sirven como orientación inicial para el proceso de búsqueda. En algunos casos es imprescindible que exista al menos un punto para definir las posiciones respectivas de cada imágen, como por ejemplo en Tarjetas Estéreo escaneadas
- Márgenes: Se mantienen los lados que no son automáticos, y se utilizan para limitar la zona de búsqueda de puntos.
- Máscaras: Se eliminan
- Textos: Se asocian todos al nuevo punto cero (pot tanto deben revisarse después)
Calidad de Puntos Automáticos#
Stmani3 utiliza el algoritmo ORB & BFMatcher de OpenCV para localizar una serie de puntos homólogos, y después selecciona entre estos puntos aquellos que:
- muestren buena correlacción (superior al 85%)
- después de alinear no tengan paralaje vertical excesivo
El proceso funciona razonablemente bien cuando las imágenes no tienen ruido ni zonas desenfocadas.
Pero incluso en fotos óptimas es frecuente que no se "vea" algún objeto próximo en la escena, con lo que estos objetos no se tienen en cuenta en el alineamiento y pueden producir violaciones de ventana. Tampoco se encuentran puntos en zonas ruidosas o desenfocadas.
Por estos motivos el Ajuste Automático de Stmani3 no siempre asegura un buen alineamiento, y es aconsejable revisarlo manualmente.
Crear Punto Manualmente#
Poner el modo de edición "Punto Nuevo", pulsando la tecla "P". Esto pone una marca en el medio de cada ventana.
Desplazar las imágenes con el ratón para definir aproximadamente la posición del punto homólogo (la marca misma no se puede mover).
Al desplazar la imagen derecha se desplaza simultaneamente la izquierda también.
Al desplazar la imagen izquierda, solo se desplaza ella.

A continuación debe realizarse el afinado del punto pulsando el botón derecho del ratón, lo que abre la Ventana de Afinado..
Ahora tenemos que colocar el punto aproximadamente en una zona donde exista suficiente contraste en las dos direcciones X Y.
A continuación se pulsa uno de los botones "Auto 10x10", "Auto 20x20" o "Auto 30x30". Los 10x10, etc son los tamaños de la ventana de correlación donde se busca la posición óptima del punto. La correlación ideal sería el 100%. Si la correlación es baja puede indicar que el par de puntos no son realmente homólogos. También ocurre cuando una la imagen no es nítida.
La ventana de correlación solo debe "ver" partes de la imagen a similar profundidad, de lo contrario en lugar de identificar un punto homólogo único, cogería una mezcla de puntos de distintos planos que producirían errores en el alineamiento. La ventana pequeña de 10x10 sirve sobre todo para determinar puntos en ramas finas o hierbas, para evitar la interferencia con objetos detrás. La ventana de 30x30 es útil sobre todo en áreas extensas cuando hay desenfoque.
Si la correlación es buena, hay que pulsar "Cargar Punto". Con esto se cierra la ventana, y el nuevo punto debe aparecer en la lista.
Modificar Punto existente#
Hay que seleccionar el Punto Homólogo a modificar, lo que se puede hacer de dos maneras:
- Pulsando la tecla "S" se entra en el modo de edición "Busca Punto". El cursor cambiará
a forma de cruz cuando se acerca a un punto, y al pulsar se selecciona el punto.
Notar que esta operación de selección de punto solo se puede realizar en la imagen derecha. - Al pulsar sobre la lista de puntos se obtiene el mismo efecto que sobre la imagen
Al seleccionar el punto cambia de aspecto en la imagen, y se remarca en la lista de puntos.

Opciones de Punto#
Pulsando el botón derecho del ratón sobre el punto seleccionado en la imagen, o sobre el mismo en la lista de puntos, aparece una ventana con opciones:
REVISAR BORRAR PONER HACER PON EN COMO ASOCIAR ASOCIAR
PUNTO TEXTO DATUM BASE TEST TÍTULO TÍTULO
Z-0.2%
- Revisar Punto: Abre una ventana de afinado que permite revisar su posición (ver Crear Punto)
- Borrar: Elimina el punto
- Poner Texto y Asociar Título: Ver apartado de edición de textos
- Hacer Datum: Asigna el punto seleccionado como Datum
- Pon en Base: Asigna el punto al plano base de un Fantograma Ver Fantogramas
- Como Test: Excluye el punto del alineamiento, pero sigue mostrando su disparidad H y V.
Márgenes#
Los márgenes de la imagen ajustada se indican con un rectángulo rojo. La posición de cada
borde es inicialmente automática, de forma que los bordes queden totalmente dentro de la futura imagen estéreo.
No obstante en ocasiones puede ser útil que los márgenes queden por fuera de ambas imágenes aunque esto suponga
que haya zonas sin cobertura estéreo. Esto se selecciona con la opción Márgenes Circumscr.[itos].
Los bordes en modo automático se indican con línea de puntos.
Pulsando la tecla "M" se entra en el modo de edición "Mueve Margen", que permite mover cada uno de los 4 bordes. El cursor cambia de forma al estar cerca de algún borde, y entonces se puede desplazar. Si el borde era automático pasa a ser manual (se indica por línea continua).
Si se desea poner el borde automático, pulsar la tecla "A" (que pone modo de edición "Margen Autom.") y pulsar sobre el borde.
Notar que estas operaciones de márgenes solo se pueden realizar en la imagen derecha.

Es posible situar los márgenes manuales por fuera de las imágenes. El tamaño de la imagen ajustada será el definido por los márgenes, y las imágenes mostrarán en negro las zonas exteriores a las imágenes:

Relación de Aspecto#
Se puede hacer que los márgenes automáticos se adapten a un factor de forma fijo (por ejemplo
16/9, 4/3, etc):
Pulsar la tecla "M" para poner el modo de edición "Mueve Margen". Hacer click-derecho sobre cualquier borde, y aparecerá
una ventana que permite poner una relación de aspecto fija.
Esto solo afecta a los bordes en modo automático.
Máscaras#
Son un complemento de los márgenes cuando se necesita ocultar alguna parte de la imagen
Es un polígono cuyos vértices se establecen y se modifican con el ratón. Está referido
al Datum al igual que los márgenes, por lo que están a la misma profundidad cero.
Para crear un polígono de máscara o modificar uno existente, pulsar la tecla "K" que pone
el modo de edición "Máscara".
Notar que las operaciones de máscaras solo se pueden realizar en la imagen derecha.
| Click en zona vacía | Crea nuevo vértice de poligonal |
| Click cerca de un vértice | Selecciona y permite desplazar el vértice |
| Click Derecho cerca de un vértice | Opciones de poligonal: Borrar vértice, Borrar todo |
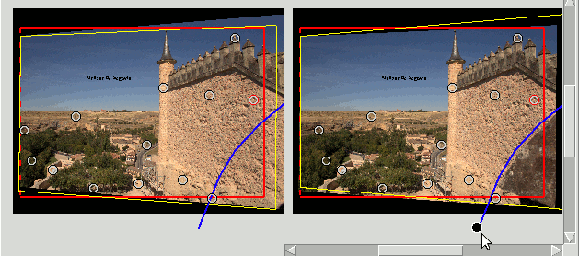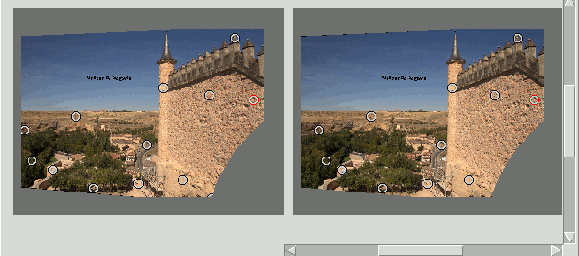
En esta imagen la máscara oculta un objeto que produce una violación de ventana:
Profundidad del Datum#
En principio los márgenes de ambas fotos se establecen con relación a uno de los puntos homólogos,
que se define como Datum. Esto implica que el punto Datum tiene disparidad cero respecto la ventana,
es decir, está a la misma profundidad que la ventana.
Pero es posible definir una profundidad diferente de cero, lo que supone crear un "Datum Virtual" por
encima o por debajo del Datum real.
Esto se hace pulsando la tecla "D" que pone el modo de edición "Datum". Pulsando entonces los botones "+", "-" y "0"
de "Profund. Datum ó Ven.Flot." se aumenta, disminuye o se pone a cero la profundidad del Datum.
La rueda de selección debajo de los botones permite elegir los incrementos de profundidad en pixels.
En estas imágenes se ha modificado la profundidad del Datum en +128 pixels. Comparar su posición (el punto rojo) con el ejemplo de "Máscaras" que tiene profundidad cero:

Ventana Flotante#
Consiste en ocultar una banda a la izquierda de la imagen izquierda, y otra a la derecha de la imagen
derecha. Esto produce la sensación de tener la ventana más cerca del observador.
El tamaño total de la imagen no se modifica, sin embargo el área útil de la imagen se reduce necesariamente.
La profundidad del Datum respecto los márgenes reales no se modifica, pero los márgenes aparentes
se observan más cerca que los reales.
La Ventana Flotante se modifica de forma similar a la profundidad del Datum, excepto que hay que pulsar la tecla "F" para poner el modo de edición "V.Flot".
En esta imagen la Profundidad del Datum es cero, pero tiene una Ventana Flotante a -128 pixels. Por tanto la disparidad del Datum respecto la ventana es idéntica al caso de Profundidad de Datum de +128 pixels. El tamaño total de imagen también es idéntico, pero la anchura útil de imagen es menor en este caso de ventana flotante debido a las bandas de ocultación:

Los siguientes LRL comparan el efecto de tener una Profundidad de Datum o tener una Ventana Flotante de profundidad equivalente. NOTA: estos pixels siempre se miden sobre las imágenes ajustadas angularmente, no sobre las renderizadas que se muestran aquí:
Prof.Datum +128 pixel: 
Vent.Flot -128 pixel: 
Orientación#
Cada imagen tiene un Botón de Orientación, que selecciona 8 posibles orientaciones:

Transformación Angular#
La fotografía estereoscópica requiere idealmente dos cámaras idénticas apuntando en paralelo, de forma que ambos sensores estén en el mismo plano. Esta es la disposición de cámaras que proporciona alineamiento perfecto, sin ningún error de disparidad vertical.
Pero los pares de fotos reales raramente cumplen esta condición. Lo que hace la Transformación Angular es
"reproyectar" las imágenes reales tomadas con dos cámaras mal apuntadas sobre un único plano de "Sensores
Virtuales", que debe ser paralelo a la línea base que une los dos lentes. Con esto se consigue el mismo
resultado que con las cámaras ideales.
Consiste matemáticamente en una transformación de Perspectiva que convierte las imágenes reales en otras
re-proyectadas. Los parámetros de transformación se calculan con el objetivo de minimizar el error de
disparidad vertical en las imágenes finales.
El siguiente gráfico muestra dos cámaras exageradamente mal apuntadas y el par de imágenes mal alineadas (en negro) que obtienen. A continuación se coloca el plano de "Sensores Virtuales" (en rojo) paralelo a la línea base que une los objetivos, y se re-proyectan las imágenes sobre este plano:
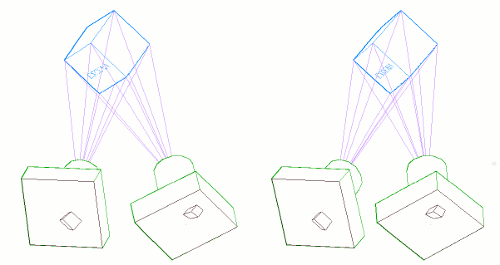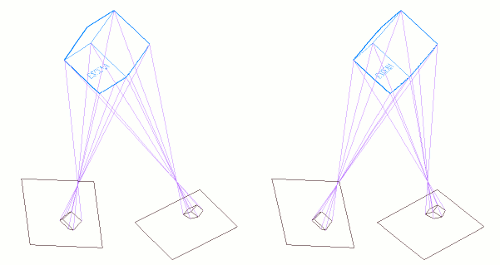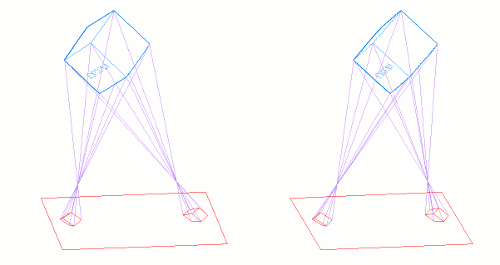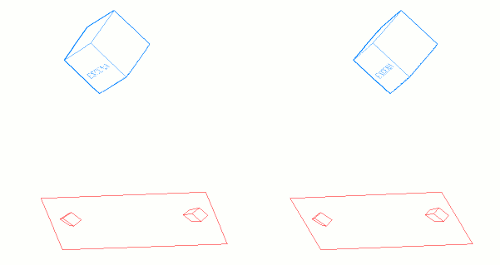
La disparidad vertical ha desaparecido en las imagenes captadas por el Sensor Virtual:
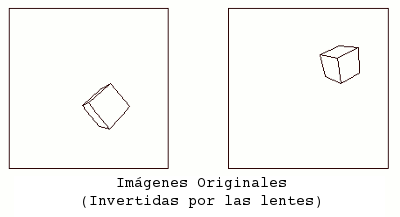

Las matemáticas que están detrás de esto se describen en este documento en ReseachGate:
- ALIGNMENT_OF_STEREO_DIGITAL_IMAGES
Parámetros de Transformación Angular#
Los parámetros de Transformación Angular se calculan automáticamente en cuanto hay puntos homólogos suficientes.
Stmani3 puede calcular un máximo de 8 parámetros de transformación, 4 de la foto izquierda y 4 de la derecha:
| Izq | Der | |
|---|---|---|
| scI | scD | Corrección de Distancia Focal Nominal |
| phI | phD | Perspectiva Horizontal (giro en el eje Y) |
| pvI | pvD | Perspectiva Vertical (giro en el eje X) |
| rtI | rtD | Rotación de Imagen (giro en el eje Z) |
Pero no todos se permiten a la vez. Por otra parte pueden usarse parámetros balanceados entre las dos fotos con un valor medio modificable por el usuario. Cada uno de los 4 parámetros básicos admite estas modalidades:
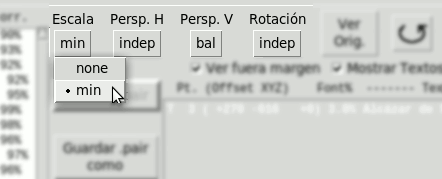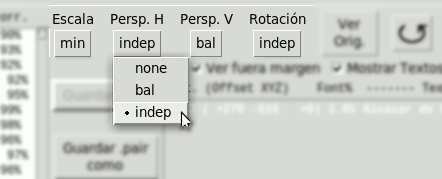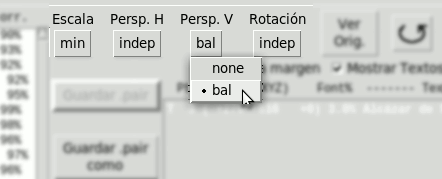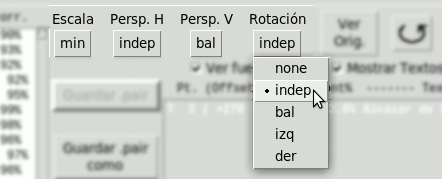
- "Escala":
- none
- min. Calcula solo un scI o scD. El que tenga el menor valor
- Perspectiva Horizontal:
- none
- bal. Calcula phD, phI como valores balanceados en torno a un punto medio ph0
ph0 es inicialmente cero, pero puede variarse manualmente. - indep. Calcula phD y phI independientemente
- Perspectiva Vertical:
- none
- bal. Calcula pvD, pvI como valores balanceados en torno a un punto medio pv0
pv0 es inicialmente cero, pero puede variarse manualmente.
- Rotación:
- none
- bal. Calcula rtD, rtI como valores balanceados en torno a un punto medio rt0
rt0 es inicialmente cero, pero puede variarse manualmente. - indep. Calcula rtD y rtI independientemente
- izq. Calcula solo rtI
- der. Calcula solo rtD
Valores medios ph0, pv0, rt0#
Los valores medios se usan en el cálculo de parámetros en modo balanceado. De hecho cualquier pulsación en los botones de control pone el permiso de ajuste correspondiente en modo bal.
Los valores medios son cero inicialmente. Se modifican manualmente pulsando la tecla "H", "V" o "R",
que ponen el modo de edición "H.Med", "V.Med" o "R.Med" respectivamente. Luego pulsando uno de los
botones bajo "Desv. Angl. H|V|R":
Los botones "+", "-" y "0" sirven respectivamente para aumentar, disminuir o poner a cero el valor medio
del ángulo correspondiente.

Resultados del Alineamiento#
Los resultados detallados se muestran en la ventana de Ajuste Angular encima de las imágenes, junto con los parámetros de las imágenes originales (ver imagen). Para cada imagen se muestran los parámetros de transformación angular (sc, ph, pv, rt) y las disparidades absolutas en pixels (c/l/v Cercana/Lejana/Vertical)
Calidad del Alineamiento#
Una indicación simple de la calidad de alineamiento se muestra al lado de cada par de miniaturas, con colores que dan una idea aproximada de la calidad.
Se proporcionan cuatro parámetros de calidad:
- "Cer." Disparidad Horizontal del punto más cercano
- "Lej." Disparidad Horizontal del punto más lejano
- "Err." Peor Disparidad Vertical
- "Corr." Peor Correlación entre izq y der de un punto homólogo
Las disparidades que muestran estos indicadores son porcentajes respecto del tamaño de pantalla del renderizado, que se define con el botón "Previsual. Render". Por lo tanto los indicadores de calidad cambian si cambia la prescripción de renderizado cambia, aunque no afecta al Alineamiento Angular y no se modifican las disparidades absolutas en pixels. El siguiente GIF muestra el efecto de cambiar entre distintas prescripciones de renderizado, observar que el indicador "Lej:" cambia entre 3.7% y 2.1%:
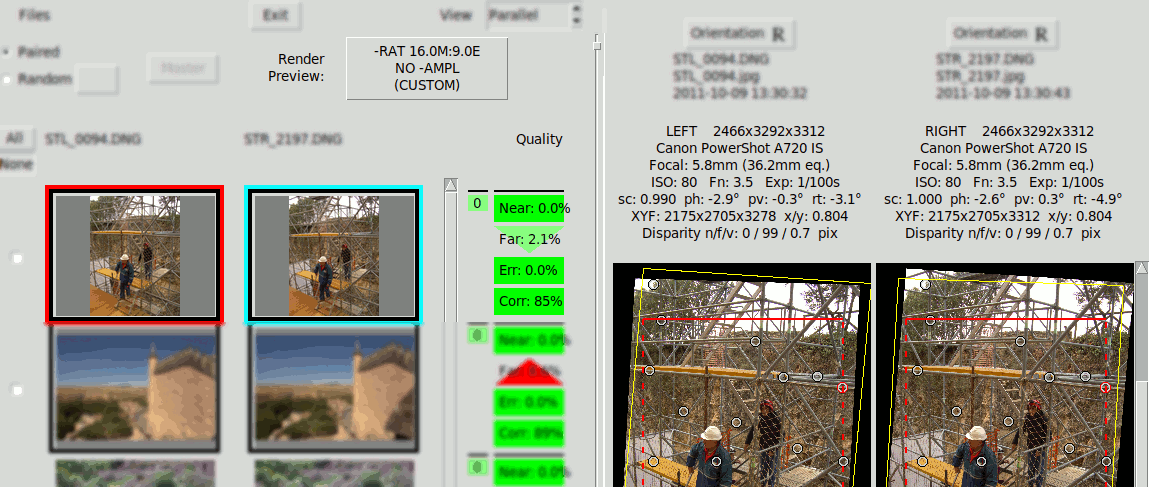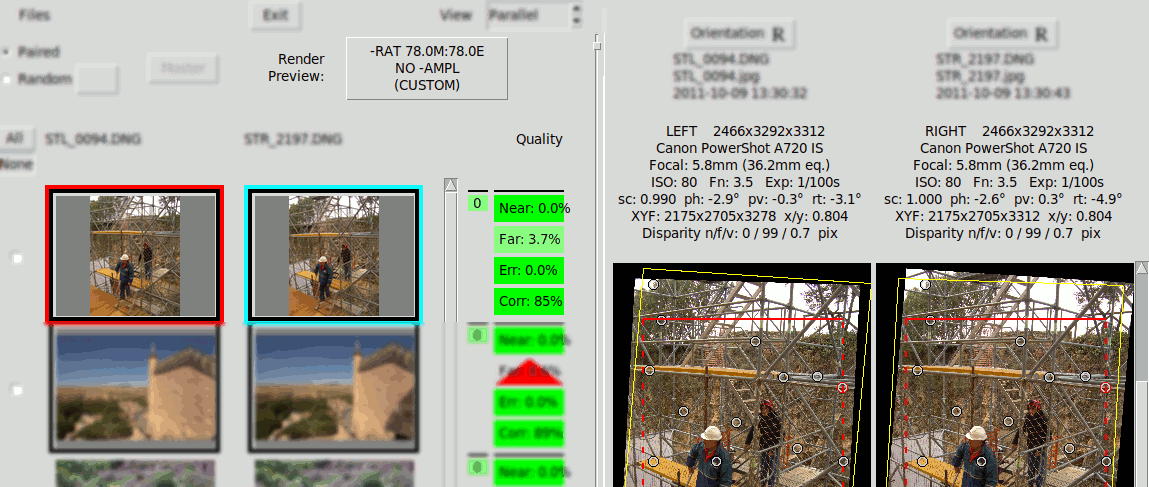
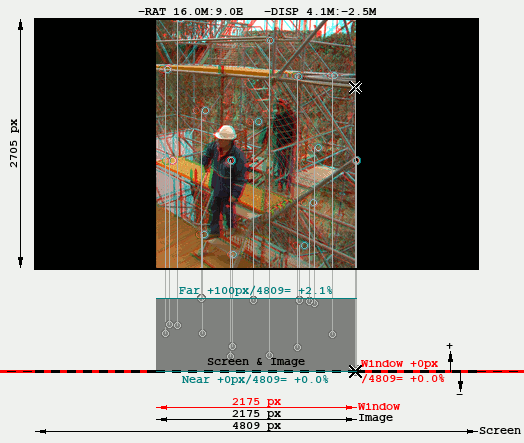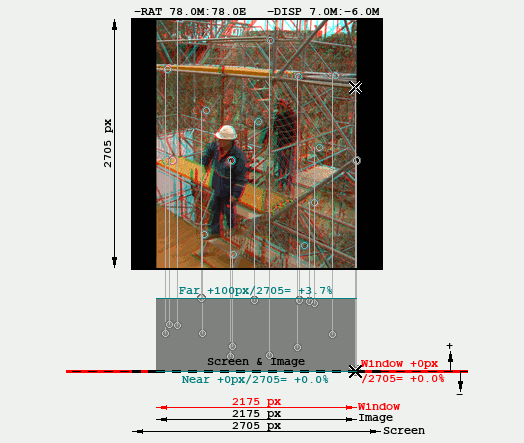
El siguiente gráfico es una especie de representación en "planta y alzado" de la misma imagen estéreo renderizada
y encajada en el Visor. La parte de abajo muestra la imagen "vista desde arriba" mostrando la disparidad
(que equivale a profundidad) de los puntos homólogos. Estos gráficos se explican en más detalle en
el capítulo de Renderizado
Se usa aquí para mostrar que la misma disparidad absoluta (100px) se percibe distinto según sea la anchura de pantalla
para la que se va a renderizar:
Pistas y Trucos#
El cálculo del Alineamiento Angular utiliza un método de mínimos cuadrados con aproximaciones sucesivas, que necesita comenzar con una estimación inicial de los parámetros. Si la estimación inicial no está muy alejada de la solución, el proceso converge rápidamente hacia la solución. Pero si está alejada el proceso puede no converger, o converger a algún mínimo local que no es solución. Puede incluso encontrar un alineamiento nominalmente bueno (con poco error vertical), pero mostrando deformaciones en la imagen que no son debidas a las condiciones de disparo de las fotos.
Stmani3 arranca suponiendo que las focales son iguales a la nominal, y que todos los ángulos son cero.
Esta suposición inicial funciona con fotos estéreo tomadas razonablemente bien:
scI = scD = 1
phI = phD = pvI = pvD = rtI = rtD = 0
Si el alineamiento no converge adecuadamente intentar lo siguiente:
- No poner los parámetros en modo "Independiente". Usar el modo "Balanceado" (o "none"), que hace más fácil la convergencia.
- Comprobar que los puntos homólogos son realmente homólogos. Comprobar en primer lugar los que tienen un valor de correlacción bajo, y/o los que tienen mayor error vertical. Comprobar siempre el Datum (a pesar de que este siempre mostrará error vertical cero).
- Asegurar que la distancia focal es correcta, al menos aproximadamente. Afecta a la transformación angular cuando los ángulos de perspectiva son grandes. Cambiarla si es necesario con Opciones de Grupo
- Modificar manualmente el valor medio de los parámetros (ver Valores Medios), y observar si la calidad del alineamiento mejora o empeora. Intentar encontrar el parámetro medio que proporciona la mejor calidad
- Algunas veces el sistema de márgenes produce inestabilidad en el alineamiento. En estos casos conviene poner todos los márgenes automáticos y con la opción Márgenes Circumscr. (ver Márgenes)
- Una vez encontrados unos valores medios que den una calidad razonable, probar a poner otra vez los parámetros en modo "Independiente". Ahora hay más posibilidades de converger debido a que los valores medios mejoran la estimación inicial.
- A veces no es fácil encontrar la estimación inicial que converja (hay algo de magia escondida:-).
Sin embargo en teoría cualquier par de fotos estéreo se puede alinear.
Con pares estéreo especialmente difíciles ayuda tener alguna idea de las condiciones de disparo: si una cámara estaba por encima de otra, o muy rotada, etc.
Fichero *.pair#
Es la salida del proceso de Ajuste Angular
Contienen todos los datos relevantes para realizar posteriormente el renderizado.
Es posible tener más de un fichero *.pair asociado a un par de imágenes. Se genera un
nuevo *.pair pulsando el botón "Salvar Como"
El ejemplo es de una imagen Fantograma, que incluye varios puntos Base y también un punto de Test:
{'FOTOS': {'der': {'hora': '2021-05-25 20:14:30',
'nombre_fich': 'DSCF7442.RAF',
'xyf': (4952, 3288, 7344.1)},
'izq': {'hora': '2021-05-25 20:14:02',
'nombre_fich': 'DSCF7441.RAF',
'xyf': (4952, 3288, 7344.1)}},
'REV': '20210714',
'TEXTOS': [{'borde': None,
'color': '#FFFFFF',
'offset': (0, 0, 0),
'pto': 6,
'texto': '',
'tfont%': 2.5}],
'TRANSF': {'AJUSTE': {'angular': {'der': {'orient': 1,
'ph': 0.0100718684,
'pv': 0.7009470301,
'rt': 0.0057924586,
'sc': 0.9784098646},
'izq': {'orient': 1,
'ph': -0.0100718684,
'pv': 0.6953163715,
'rt': 0.0117340792,
'sc': 1.0},
'pbx': -0.0052229,
'pby': -0.0003711,
'pvo': 0.0},
'margenes': {'desv_z': 0,
'float': 0,
'margen_ratio': None,
'origen': (-2008, -2227),
'parche': [],
'pto': 6,
'tamanyo': (3040, 2932)},
'permisos': {'auto_margen': {'bottom_side': False,
'circumscribed': False,
'left_side': False,
'right_side': False,
'top_side': True},
'ph': 'bal',
'phg': 'bal',
'pv': 'bal',
'rt': 'bal',
'sc': 'min'},
'ptos_homologos': [{'der': (1271.9, 253.2),
'especial': 'B',
'izq': (1598.7, 284.6)},
{'der': (1100.3, 1934.0),
'especial': 'B',
'izq': (1486.9, 1965.9)},
{'der': (1235.9, 1334.7),
'especial': 'B',
'izq': (1602.0, 1366.8)},
{'der': (2503.5, 329.9),
'especial': 'T',
'izq': (2835.0, 386.2)},
{'der': (2539.9, 900.4),
'especial': 'B',
'izq': (2892.1, 955.9)},
{'der': (2084.0, 2085.1),
'especial': 'B',
'izq': (2480.1, 2133.7)},
{'der': (3187.1, 2086.1),
'especial': 'B',
'izq': (3583.3, 2153.2)},
{'der': (3942.6, 818.3),
'especial': 'B',
'izq': (4288.2, 900.4)},
{'der': (4136.7, 1951.7),
'especial': 'B',
'izq': (4523.1, 2035.1)},
{'der': (2842.0, 1509.0),
'especial': None,
'izq': (3241.0, 1571.2)},
{'der': (2105.0, 503.0),
'especial': None,
'izq': (2478.1, 551.7)},
{'der': (1553.1, 1294.1),
'especial': None,
'izq': (1962.6, 1333.2)},
{'der': (3270.9, 456.6),
'especial': None,
'izq': (3645.9, 528.7)},
{'der': (3721.4, 329.1),
'especial': 'B',
'izq': (4048.2, 407.8)},
{'der': (3000.0, 2776.0),
'especial': None,
'izq': (3265.6, 2838.3)},
{'der': (1037.9, 2762.9),
'especial': None,
'izq': (1261.6, 2792.9)},
{'der': (700.0, 993.0),
'especial': None,
'izq': (898.9, 1011.9)}]},
'PIL': {'tupla_inv': {'der': (1.1478265063,
-0.2524612779,
726.9477250894,
-0.0052690051,
0.7216380647,
19.9248746777,
-1.755e-06,
-9.95138e-05),
'izq': (1.1543832346,
-0.2588393275,
1042.4926653797,
0.0179052593,
0.7169832364,
40.2617744533,
3.4e-08,
-0.000100186)}},
'VALOR': {'corr_min%': 79.0,
'prl_cercano': -33.3,
'prl_lejano': 122.0,
'prl_vertical': 1.3}}}